La nuova frontiera dell'RNA interference
7 novembre 2006
E’ di queste settimane la notizia del premio Nobel per la medicina assegnato ai genetisti americani Andrew Z. Fire e Craig C. Mello per la scoperta del meccanismo dell’RNA interference o “interferenza”.
In realtà occorre precisare che le prime evidenze sull’RNAi furono identificate per caso in seguito ad esperimenti fatti sul fiore di Petunia (co-soppressione dell'espressione genica) e sui virus (silenziamento genico indotto da virus) a cavallo tra gli anni 80 e gli anni 90. I fenomeni individuati fino a questo punto furono denominati silenziamento genico post-trascrizionale.
Il termine RNAi è stato inizialmente coniato dai due Autori nel 1998 per spiegare l’osservazione che l’RNA a doppio filamento (dsRNA) può bloccare l’espressione genica quando introdotto nei vermi nematodi. L’RNAi è stato da allora scoperto in un largo numero di organismi, incluse le mosche, i tripanosomi la planaria, l’ hydra , il pesce zebra e il topo e sembra essere collegato al fenomeno del silenziamento genico nelle piante e nel fungo Neurospora (detto “quelling”).
L’ ”RNA interference” interviene a livello post-trascrizionale e coinvolge la degradazione dell’RNA messaggero. Esso rappresenta un meccanismo di difesa cellulare evolutivamente conservato per controllare l’espressione di geni esogeni nella gran parte degli eucarioti incluso l’uomo; negli animali potrebbe giocare un ruolo importante nella protezione del genoma contro l’instabilità causata dall’accumulo di trasposoni e di sequenze ripetitive. Il meccanismo dell’interferenza è innescato da RNA a doppio filamento (dsRNA) che causa la degradazione sequenza-specifica di un mRNA bersaglio omologo a singolo filamento. I mediatori della degradazione dell’mRNA sono dei piccoli duplex a RNA o “small interfering RNA” (siRNA) i quali sono prodotti da un lungo RNA a doppio filamento tramite un taglio enzimatico nella cellula.
Il mio progetto di ricerca, che sto svolgendo nel laboratorio del prof. Sergio Pimpinelli (presso l’Istituto di Genetica dell’Università “La Sapienza” di Roma), si prefigge l’obiettivo di identificare e caratterizzare geni coinvolti nell’ RNAi sfruttando un sistema presente in uno degli organismi modello più utilizzati in biologia, il moscerino della frutta Drosophila melanogaster; questo sistema, chiamato crystal-Stellate, permette di identificare geni coinvolti nell’RNAi tramite l’osservazione di un fenotipo presente nelle cellule della linea maschile del moscerino. I geni mutanti per l’RNAi determinano la produzione e l’epressione di una proteina chiamata STELLATE che produce cristalli negli spermatociti primari e negli spermatidi dei moscerini maschi. La forma dei cristalli può essere ad ago o a stella a seconda dell’ allele ste.
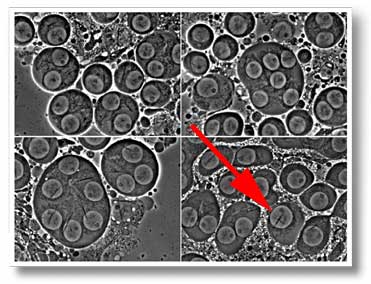 In condizione di default, un locus eterocromatico sul cromosoma Y (il locus crystal) reprime un locus eucromatico sul cromosoma X (il locus Stellate); i loci Ste e cry sono composti di sequenze omologhe ripetitive. Il silenziamento operato da crystal sulle sequenze di Stellate sembra coinvolgere un meccanismo di RNA interference (RNAi). Quando l’ RNAi non funziona, le sequenze Stellate vengono trascritte e tradotte e la proteina STELLATE viene espressa. La produzione di questa proteina, oltre a determinare la presenza dei cristalli, porta ad una serie di difetti meiotici che in ultimo causano sterilità.
In condizione di default, un locus eterocromatico sul cromosoma Y (il locus crystal) reprime un locus eucromatico sul cromosoma X (il locus Stellate); i loci Ste e cry sono composti di sequenze omologhe ripetitive. Il silenziamento operato da crystal sulle sequenze di Stellate sembra coinvolgere un meccanismo di RNA interference (RNAi). Quando l’ RNAi non funziona, le sequenze Stellate vengono trascritte e tradotte e la proteina STELLATE viene espressa. La produzione di questa proteina, oltre a determinare la presenza dei cristalli, porta ad una serie di difetti meiotici che in ultimo causano sterilità.
Attraverso una semplice osservazione dei cristalli presenti nelle cellule, si possono quindi identificare tutti quei geni potenzialmente coinvolti nell’interferenza. Una volta identificati, questi geni vengono caratterizzati, clonati e si cerca di capirne il ruolo e il funzionamento nel macchinario dell’ RNAi.
Degno di nota è il fatto che Drosophila è l’unico organismo presente in natura in cui sia possibile osservare l’RNAi “in vivo”.
Le potenzialità del meccanismo dell’interferenza sono enormi; l’RNAi ha diverse applicazioni nel campo dell’ingegneria genetica. In particolare viene utilizzato per silenziare l’espressione genica di qualunque gene. Attraverso lo spegnimento di un determinato gene, è possibile identificarne il ruolo. Lo spegnimento mediato da RNAi è genericamente definito knock-down.
Tra i vantaggi di questa tecnica, c’è anche quello di poter ripristinare l’attività del gene silenziato. L’RNAds che avvia l’RNAi può anche essere utilizzato come una sorta di “farmaco”, in particolare nell’ambito della terapia genica.
Un ulteriore vantaggio dell’RNAi è il fatto che esso agisce solo su molecole di acido nucleico già trascritte; le terapie a base di RNAi quindi non solleverebbero tutti i dubbi etici che attualmente qualsiasi tipo di intervento sul DNA comporta.
© Maria Cristina Onorati
Glossario:
• eterocromatico: dell’ eterocromatina; l’eterocromatina indica quelle regioni cromosomiche che rimangono in uno stato condensato durante la gran parte del ciclo cellulare. È distinta in due tipi, facoltativa e costitutiva.
• eucromatico: dell’eucromatina; l’eucromatina contiene la gran parte dei geni strutturali.

